最近拿到了SEER的癌症发病数据,准备做一些流行病学的分析。但是SEER提供的数据是经过编码的,如果不使用他们定制的分析软件就只能先自己转码后再进行分析。我还是更喜欢用R一些,所以果断选择后者。
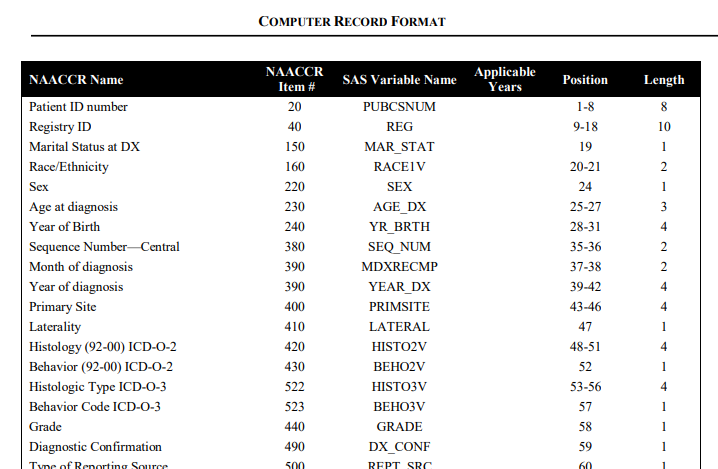
这套数据编码后能压缩掉不少空间,但是要分析的时候也挺麻烦的,由于是根据position来确定变量数值,因此解码就是把这些变量按position和length信息分隔开来,详细的编码情况如下:
然后我就根据编码规则在写了如下的R代码:
#install.packages('stringr')
library(stringr)
raw_data <- readLines(
'~/SSA-LungCancer/datasets/SEER_1975_2016_TEXTDATA/incidence/yr1975_2016.seer9/RESPIR.TXT')
transcode_data <- setNames(data.frame(matrix(ncol = 143, nrow = 0)), c("PUBCSNUM", "REG",
"YR_BRTH", "SEQ_NUM", "MDXRECMP", "YEAR_DX", "PRIMSITE", "LATERAL",
"HISTO2V", "BEHO2V", "HISTO3V", "BEHO3V", "GRADE", "DX_CONF",
#此处省略100+变量
"TNMEDNUM", "METSDXLN", "METSDXO"))
for(i in c(1:length(raw_data))){
transcode_data[i,1] <- str_sub(raw_data[i], start = 1,end = 8)
transcode_data[i,2] <- str_sub(raw_data[i], start = 9,end = 18)
transcode_data[i,3] <- str_sub(raw_data[i], start = 19,end = 19)
#此处省略100+行
print(paste("Row", i, "Transcoded"))
}
write.csv(transcode_data,file = "~/SSA-LungCancer/processed_data/seer_1975_2016_respir_incidence.csv")
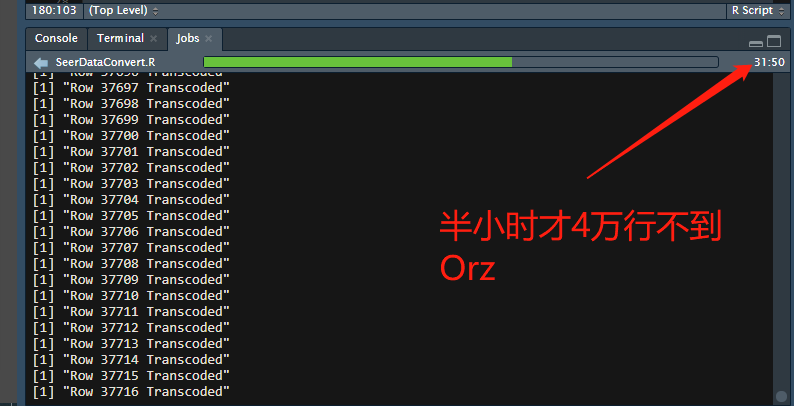
然后就跑呀!在云筏的R云服务器中跑的,他们的服务器CPU主频是3.6GHz,应该是目前云厂商中最高的了(阿里云的多为2.6GHz),比我2.2GHz的笔记本好太多。但是....
实在是太慢了,半小时才跑了十分之一,不能忍!我反省!
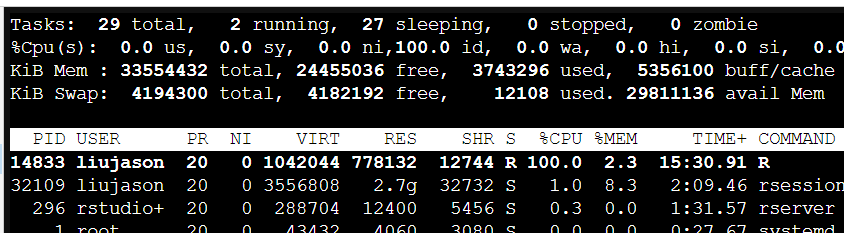
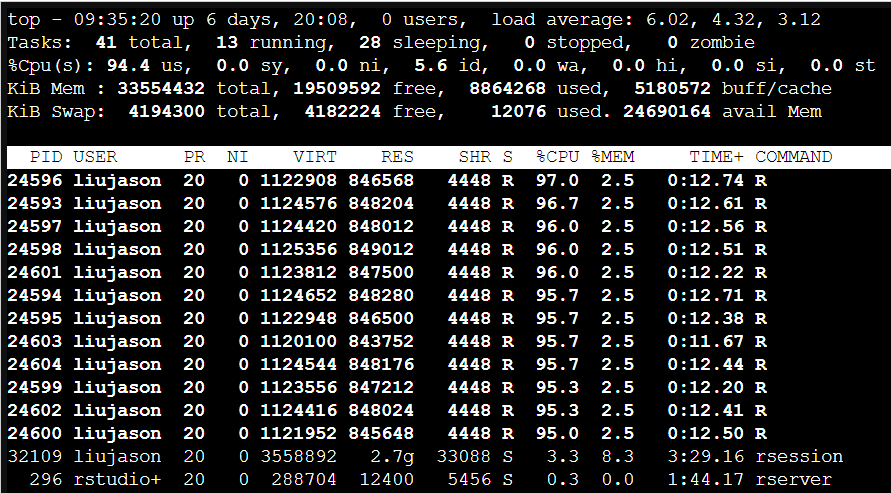
的确是我的代码写的有问题,为什么要用循环分割的方法呢?然而我也没想到什么更好的算法,那就从算力角度来看看怎么加速吧。于是进terminal用top看看:
emm...单核跑满了,众所周知for循环是要一个个来的,因此也只能单线程计算。那么就简单了,这个数据的拆分工作是相互独立的,我们用多线程呗~
使用到的R包:parallel/doParallel/foreach
代码如下:
#install.packages('stringr', 'doParallel')
library(doParallel)
library(stringr)
#read data
raw_data <- readLines(
'~/SSA-LungCancer/datasets/SEER_1975_2016_TEXTDATA/incidence/yr1975_2016.seer9/RESPIR.TXT')
#define dataframe
transcode_data <- setNames(data.frame(matrix(ncol = 143, nrow = 0)), c("PUBCSNUM", "REG",
"YR_BRTH", "SEQ_NUM", "MDXRECMP", "YEAR_DX", "PRIMSITE", "LATERAL",
"HISTO2V", "BEHO2V", "HISTO3V", "BEHO3V", "GRADE", "DX_CONF",
#此处省略100+变量
"TNMEDNUM", "METSDXLN", "METSDXO"))
#configure multi-threading
numCores <- detectCores()
registerDoParallel(numCores)
#transcode
foreach(i = 1:length(raw_data)) %dopar% {
transcode_data[i,1] <- str_sub(raw_data[i], start = 1,end = 8)
transcode_data[i,2] <- str_sub(raw_data[i], start = 9,end = 18)
transcode_data[i,3] <- str_sub(raw_data[i], start = 19,end = 19)
#此处省略100+行
print(paste("Row", i, "Transcoded"))
}
#write to file
write.csv(transcode_data,file = "~/SSA-LungCancer/processed_data/seer_1975_2016_respir_incidence.csv")
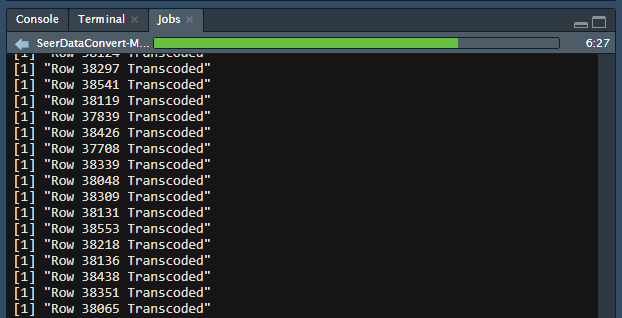
写好后立刻试一下速度,大幅提升速度妥妥的!
再来看一下线程的使用情况:
这时候有同学就要问了,明明是用了12个线程,为什么没有得到12倍的效果呢?那是因为单线程的时候CPU自动睿频了,云筏的R服务器真的是良心呀!
以为这样就结束了吗!并没有!我~还~要~更~快~
我发现到了后面数据框的输入越来越慢,目测是因为数据量太大造成的内存问题,于是我又改进了一下代码:不追加到数据框中,直接写入文件尾部。
write.table(matrix(as.character(transcode_data), nrow=1), sep=",",
file = "~/SSA-LungCancer/processed_data/seer_1975_2016_respir_incidence.txt",
append = T, row.names=FALSE, col.names=FALSE)
实际上也就是改了两个部分,其中最主要的还是追加写入文件尾部。这样速度快了很多,实际测试下来66万行数据的录入只花了6分钟不到,再次提速!